wslにrstudio serverを入れる
下記のブログ記事を参考にインストールする.
下記を参考にインストールする.
https://cran.r-project.org/bin/linux/ubuntu/README.html
sudo apt-get update sudo apt-get install r-base
このままだとRのバージョンが古いので,下記を参考に最新のRをインストールする.
- Rstuio Serverのインストール
下記を参考にインストールする.
sudo apt install gdebi-core wget https://download2.rstudio.org/server/bionic/amd64/rstudio-server-1.2.5042-amd64.deb sudo gdebi rstudio-server-1.2.5042-amd64.deb
あとは下記コマンドでインストールされたか確認する.
rstuio-server or localhost:8787
xml2パッケージのインストールでこけた場合は下記を実行したうえで,再度インストールをする.
Sys.setenv(R_INSTALL_STAGED = FALSE)
rstudio-serverがインストール出来たら, windows側のフォルダにアクセスしやすいように下記の記事に従って設定を行う.
rstuio-serverでplotしたときに日本語が文字化けするようであれば,フォントを入れる.
sudo apt-get install fonts-ipafont
フルマラソンの完走タイム
あまりの運動不足から最近ランニングをはじめ、継続するための目標としてフルマラソンを走ってみたいと思っている。 フルマラソンをとても走れるとは思えなかったが、調べると意外と走れるようである。 走れるといってもそう簡単にできるものではないので、下調べをしていると 自分がどの程度で走れるのか、また、ほかの人がどの程度で走っているのかに興味が出てきた。
スポロクリザルトというサイトで一部マラソン大会の結果が公表されていたので、 一般市民が参加しそうで比較的有名そうな金沢マラソンの記録をもとに、マラソン大会出場者の完走タイムについて簡単に調べてみた。
# パッケージの読込 library(tidyverse) library(rvest) library(lubridate)
# データの取得 ## 男子総合の結果を順次取得する ## 時間もかかるため、ここでは読み込んだ既に取得したデータを読み込むことにする # # 男子 # adress = "https://sporoku.jp/result/kanazawa_20191027/list?race_id=696&div_id=584&page=" # pages = 1:209 # # # dt_male = list() # for(i in pages){ # dt_male[i] = # read_html(paste0(adress, pages[i])) %>% # html_table() # } # saveRDS(dt_male,"kanazawa_marathon_male.rds") # # # 女子 # adress = "https://sporoku.jp/result/kanazawa_20191027/list?race_id=696&div_id=585&page=" # pages = 1:67 # # dt_female = list() # for(i in pages){ # dt_female[i] = # read_html(paste0(adress, pages[i])) %>% # html_table() # } # saveRDS(dt_female,"kanazawa_marathon_female.rds") dt_male = readRDS("kanazawa_marathon_male.rds") dt_female = readRDS("kanazawa_marathon_female.rds") data = map(list(dt_male, dt_female), ~ do.call("rbind",.x)) %>% map(~ .x[,1:4] %>% as_tibble() %>% mutate(time = lubridate::hms(タイム), ranking = str_replace(順位,"位","") %>% as.integer())) %>% map2(c("male", "female"), ~ mutate(.x, gender = .y)) data = do.call("rbind",data) data = group_by(data, gender) %>% mutate(ranking_percentile = ranking/max(ranking)) %>% ungroup()
# 順位パーセンタイル値と完走時間の関係をプロット data %>% ggplot(aes(ranking_percentile, time, color = gender))+ geom_point()+ scale_y_time()
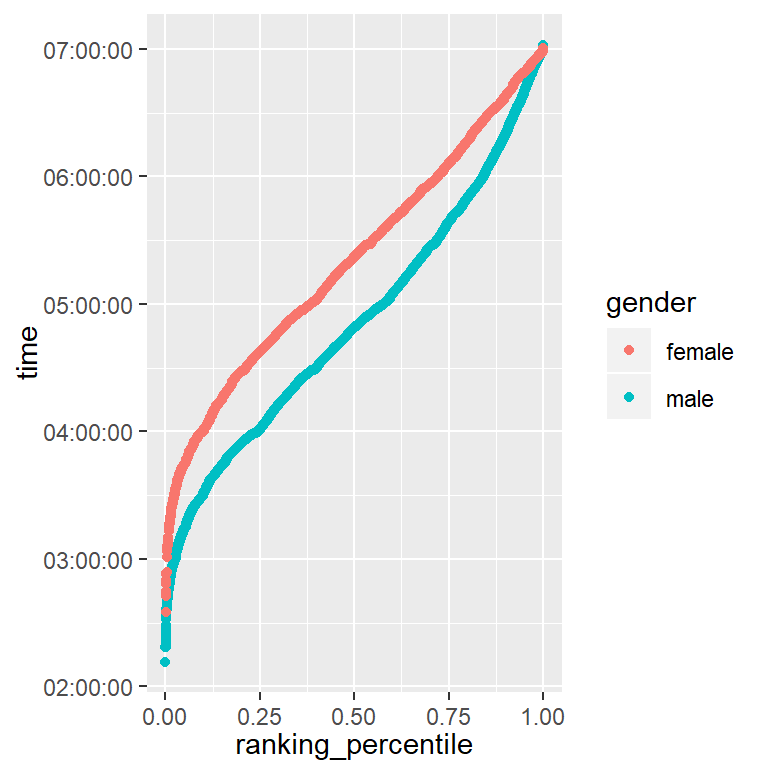
4人に1人はサブ4を達成していることが分かる。 このままでは詳細が見ずらいので、順位のパーセンタイル値ごとに完走時刻をプロットする。
data %>% mutate(percentile_factor = cut(ranking_percentile,breaks = seq(0,1,by=.1))) %>% ggplot(aes(ranking_percentile, time, color=gender))+ geom_point()+ scale_y_time() + scale_x_continuous(breaks=seq(0,1,by=.1))+ facet_wrap(~percentile_factor, scales = "free")

サブ3.5を達成すると上位10%に入ることができるようである。
また、サブ4, サブ5近辺でグラフの傾きが少し変わっていることが確認できる。
これは、それぞれの時間内で完走を目指すためだろう。
## 完走時間のパーセンタイル値 data %>% select(time, gender) %>% group_split(gender) %>% map(~ pull(.x,time) %>% as.numeric() %>% quantile(seq(0,1,by=.05)) %>% data_frame(percentage = names(.),value=.) %>% mutate(h = floor(value/3600), m = floor((value-h*3600)/60), s = floor(value-h*3600-m*60)) %>% mutate(time = paste(h,m,s, sep = ":") %>% hms()) %>% select(percentage, time)) %>% map2(.y=c("female","male"),~mutate(.x,gender=.y)) %>% do.call(what="rbind",args=.) %>% spread(gender, time, convert=T) %>% mutate(num=str_extract(percentage,"\\d+") %>% as.integer()) %>% arrange(num) %>% select(-num) %>% mutate_at(vars(ends_with("male")), hms) %>% mutate(diff_time = as.duration(female - male)) %>% knitr::kable(format="markdown")
| percentage | female | male | diff_time |
|---|---|---|---|
| 0% | 2H 34M 52S | 2H 11M 36S | 1396s (~23.27 minutes) |
| 5% | 3H 44M 25S | 3H 14M 22S | 1803s (~30.05 minutes) |
| 10% | 4H 0M 18S | 3H 30M 22S | 1796s (~29.93 minutes) |
| 15% | 4H 15M 23S | 3H 44M 18S | 1865s (~31.08 minutes) |
| 20% | 4H 28M 6S | 3H 54M 24S | 2022s (~33.7 minutes) |
| 25% | 4H 37M 35S | 4H 1M 3S | 2192s (~36.53 minutes) |
| 30% | 4H 46M 56S | 4H 12M 57S | 2039s (~33.98 minutes) |
| 35% | 4H 55M 21S | 4H 22M 52S | 1949s (~32.48 minutes) |
| 40% | 5H 1M 54S | 4H 29M 53S | 1921s (~32.02 minutes) |
| 45% | 5H 13M 17S | 4H 39M 50S | 2007s (~33.45 minutes) |
| 50% | 5H 22M 27S | 4H 48M 48S | 2019s (~33.65 minutes) |
| 55% | 5H 30M 23S | 4H 56M 20S | 2043s (~34.05 minutes) |
| 60% | 5H 39M 5S | 5H 4M 27S | 2078s (~34.63 minutes) |
| 65% | 5H 47M 47S | 5H 15M 21S | 1946s (~32.43 minutes) |
| 70% | 5H 56M 13S | 5H 26M 49S | 1764s (~29.4 minutes) |
| 75% | 6H 6M 0S | 5H 38M 33S | 1647s (~27.45 minutes) |
| 80% | 6H 16M 25S | 5H 49M 52S | 1593s (~26.55 minutes) |
| 85% | 6H 28M 3S | 6H 2M 59S | 1504s (~25.07 minutes) |
| 90% | 6H 37M 51S | 6H 19M 38S | 1093s (~18.22 minutes) |
| 95% | 6H 49M 7S | 6H 39M 38S | 569s (~9.48 minutes) |
| 100% | 7H 0M 19S | 7H 1M 55S | 96s (~1.6 minutes) |
上記の結果から、男子のタイムに30分を加えたタイムが、同じ走力を持つ女子と考えられる。
mapviewで躓いたので備忘録
mapviewの躓いた点の備忘録
久しぶりにmapviewパッケージをを触ったら躓いたので、メモ。
利用するライブラリ
library(tmap) library(leaflet) library(mapview)
利用するデータ
data("franconia") class(franconia)
[1] "sf" "data.frame"
描画対象物の色を白系にした場合に描画できない問題
描画対象物を白色を指定した場合に、地図が表示できなくなる場合は、以下のようにmap.types=にベースとなるタイルを指定してやるのが良い。 map.typesにはサイトから好みの名前を指定する。
mapview(franconia, map.types="OpenStreetMap.Mapnik", col.regions="snow")
tmapを利用した背景地図付きの地図の保存方法
下記だと描画モード(Rstudio上)では表示されるが、画像ファイルとして保存する際には背景画像が消えてしまう。
tmap_mode("view") m = tm_shape(franconia)+ tm_polygons() m
# 下記では背景地図が保存されない tmap_save(m, "test.png")
このように背景地図が描画されない
解決方法としてはmapshot()関するを利用する。
mapshot(tmap_leaflet(m), file="test2.png")
 表示できた!!!
表示できた!!!
nestしたデータフレームの扱い
nestしたデータフレームを扱い方(メモ用)
library(tidyverse) iris %>% group_by(Species) %>% nest() %>% mutate(data = map2(data, Species, ~mutate(.x, Species_index = as.numeric(.y)))) %>% mutate(id = seq(1,3)) %>% mutate(data = map2(data, id, ~slice(.x, seq.int(.y)))) %>% mutate(MeanSepalLength = map_dbl(data, ~pull(.x, Sepal.Length) %>% mean))
上記の結果以下のデータを得ることができる。
Species data id MeanSepalLength <fct> <list> <int> <dbl> 1 setosa <tibble [1 x 5]> 1 5.1 2 versicolor <tibble [2 x 5]> 2 6.7 3 virginica <tibble [3 x 5]> 3 6.40
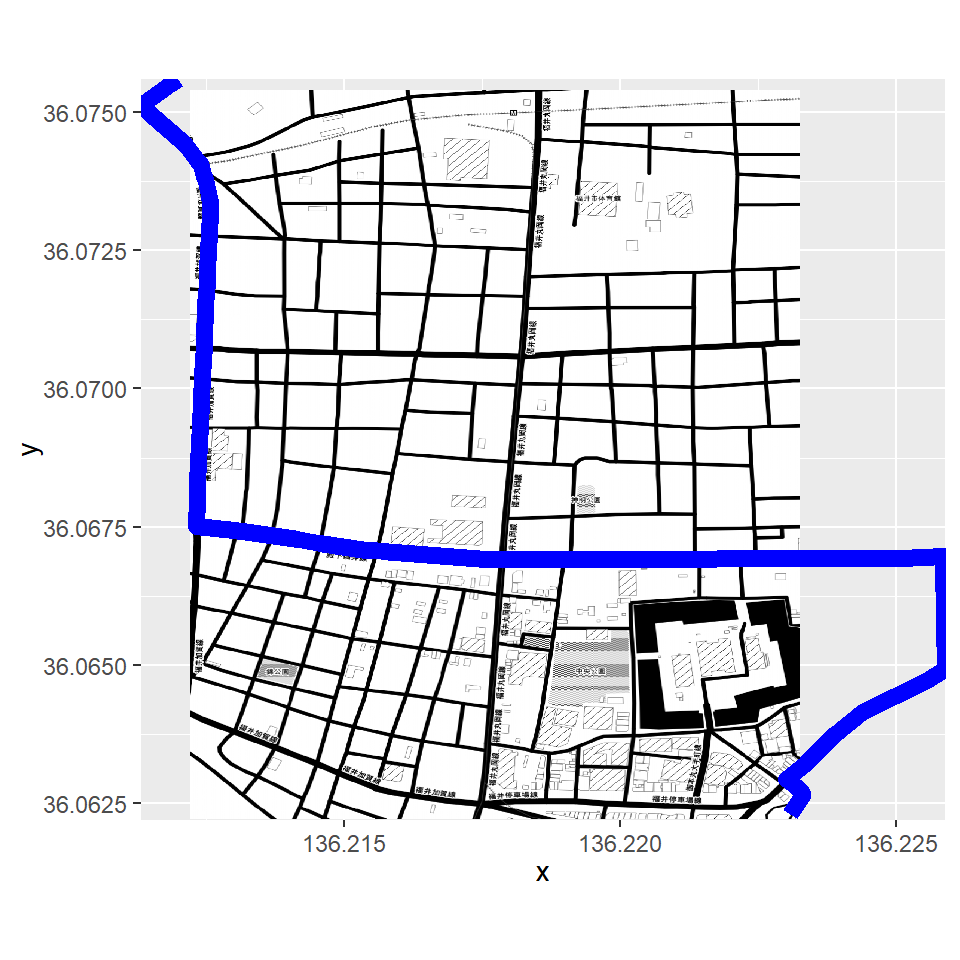
ルート探索
# 必要なライブラリのロード library(tidyverse) library(sf) library(mapview) library(stplanr) library(OpenStreetMap) # 2地点間のポイントデータ作成 sfd = tribble(~lat,~lon,~location, 36.075411, 136.212213,"福井大学", 36.062244, 136.223214, "福井駅") %>% st_as_sf(coords = c("lon", "lat"), crs = 6668) # linestringへ変換 sf_line = sfd %>% st_combine() %>% st_cast("LINESTRING") %>% st_sf(a = 1) # ラインからルートデータへの変換 line2route(sf_line, "route_osrm") %>% mapview()
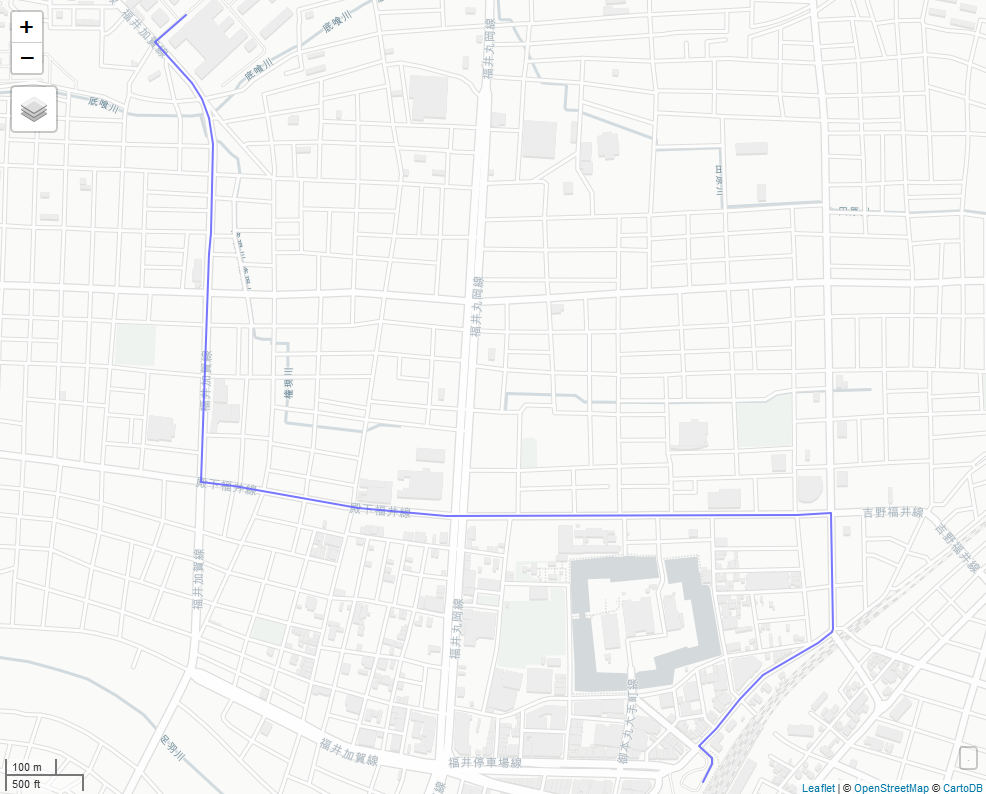
library(ggspatial) sf_route = line2route(sf_line, "route_osrm") ggplot(sf_route) + annotation_map_tile(zoomin = 0) + geom_sf()
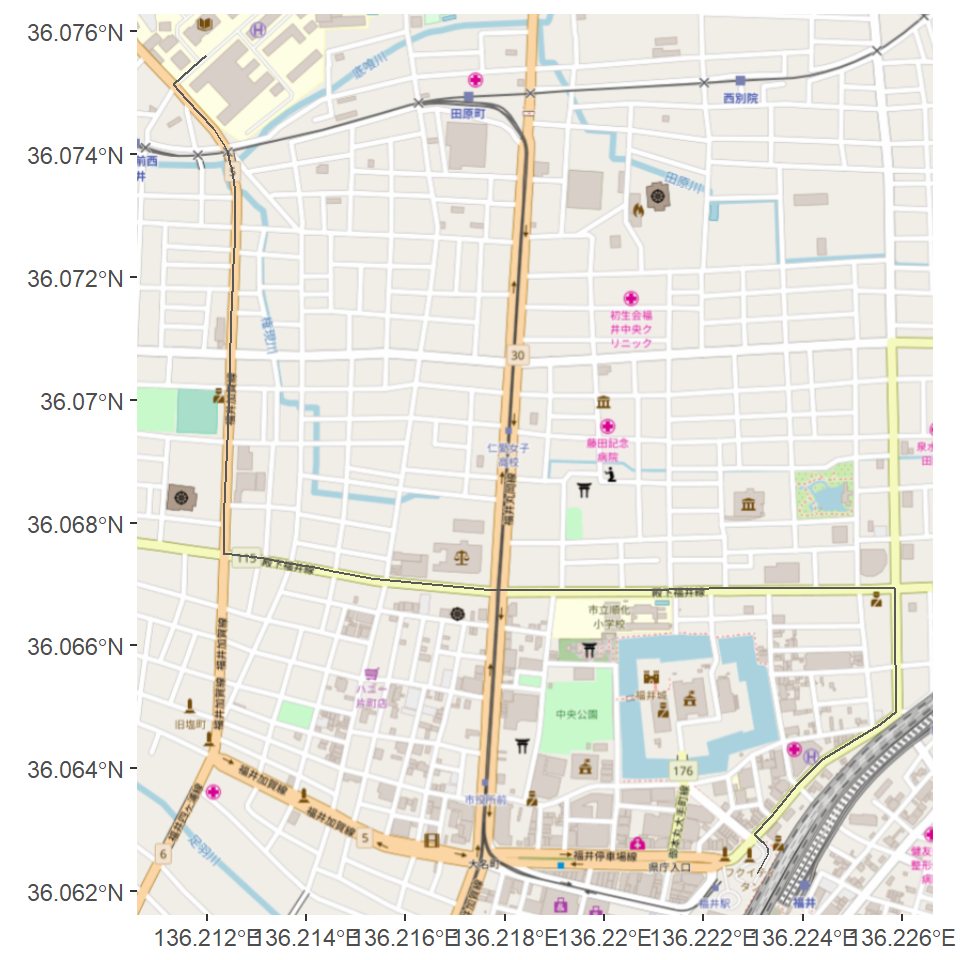
upperLeft = st_bbox(sf_route) %>% as.vector() %>% .[c(2, 1)] lowerRight = st_bbox(sf_route) %>% as.vector() %>% .[c(4, 3)] osmap = OpenStreetMap::openmap(upperLeft = upperLeft, lowerRight = lowerRight, type = "stamen-toner", mergeTiles = T) osmaplocation = OpenStreetMap::openproj(osmap, st_crs(sfd)$proj4string)
autoplot(osmaplocation) + geom_path(data = st_coordinates(sf_route)[,1:2] %>% as.data.frame() %>% `colnames<-`(c("x", "y")), color="blue", size = 3)
GeoPackageが便利
GISで利用するデータのファイル形式について、 これまでいろいろな不満がありながらも、 そのほかの代替手段を知らずやむを得ずシェープファイルを利用していた。
たまたま、sfパッケージの入出力 について調べていたら、GeoPackageというファイル形式があること知った。
少し調べたところ、最近普及し始めているようなのでメモ。
参考としたWebSite
GeoPackageでポータブルOSMは約2年前(2016年)に書かれた記事である。 シェープファイルを利用していて常に悩まされていることが書かれており、 大体のファイル形式としてGeoPackageが挙げられている。
以下のような悩みが解決されるらしい。
- 文字コードによる文字化け
- ファイルが1つ(.gpkg)だけ
- 列名の文字数
既にQGIS、ArcGIS、GDAL/OGRで利用できるらしい。 また、QGIS3.0からは保存の際のデフォルトのファイル形式になるらしい。 ということで、全然動向を知らなかったが、GeoPackageに移行する時期に 既になっているらしいので、どんどん使っていこうと思う。
sf vignette 6
Rのsfパッケージの vignette6について、 これまでのvignetteで書けなかったことなどを雑多に書いてあるので、 あまりまとまりがない感じである。
EPSGとは
EPSGは空間座標参照システムとしてよく知られており、 IOGP (International Association of Oil & Gass Producers)によってメンテナンスされている。 下記の関数を利用することでEPSG一覧を見ることができる。
rgdal::make_EPSG()
sfで第二のgeometry columnを扱うには
sfオブジェクトは一つ以上のgeometry list-columnを持つことができるが、有効となるのは一つだけであり、
st_geometry()関数によって取得できる。
printメソッドを利用することでgeometry columnが有効か確認することができる。
library(sf) demo(nc, ask =F, echo = F)
nc$geom2 = st_centroid(st_geometry(nc)) print(nc, n = 2)
Simple feature collection with 100 features and 14 fields
Attribute-geometry relationship: 0 constant, 8 aggregate, 6 identity, 1 NA's
Active geometry column: geom
geometry type: MULTIPOLYGON
dimension: XY
bbox: xmin: -84.32385 ymin: 33.88199 xmax: -75.45698 ymax: 36.58965
epsg (SRID): 4267
proj4string: +proj=longlat +datum=NAD27 +no_defs
First 2 features:
AREA PERIMETER CNTY_ CNTY_ID NAME FIPS FIPSNO CRESS_ID BIR74
1 0.114 1.442 1825 1825 Ashe 37009 37009 5 1091
2 0.061 1.231 1827 1827 Alleghany 37005 37005 3 487
SID74 NWBIR74 BIR79 SID79 NWBIR79 geom
1 1 10 1364 0 19 MULTIPOLYGON (((-81.47276 3...
2 0 10 542 3 12 MULTIPOLYGON (((-81.23989 3...
geom2
1 POINT (-81.49826 36.4314)
2 POINT (-81.12515 36.49101)
geometry columnを変更するにはst_geometry<-またはst_set_geometry関数を利用する。
plot(st_geometry(nc))
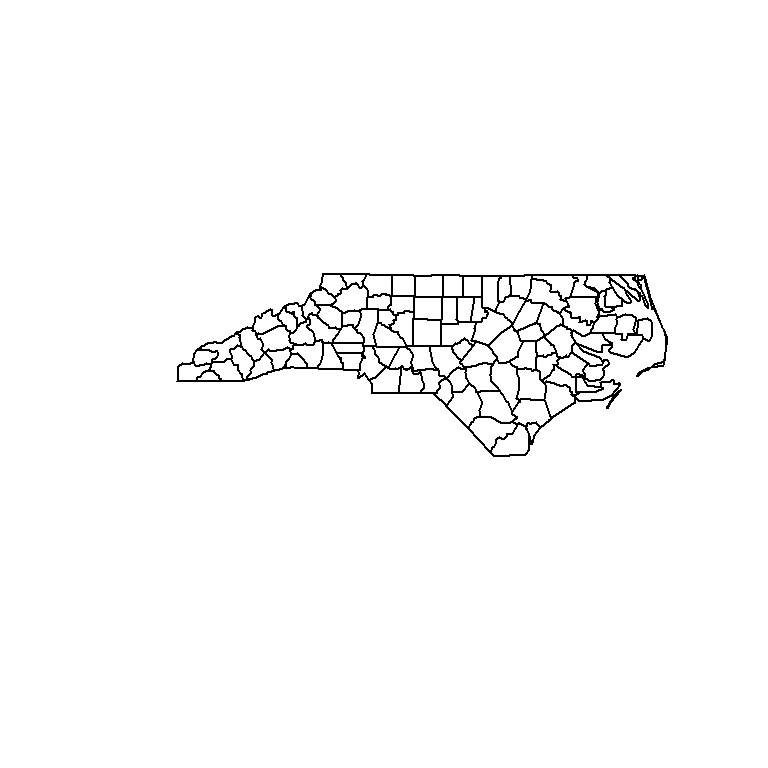
st_geometry(nc) <- "geom2" plot(st_geometry(nc))

st_simplifyでのトポロジー関係の維持について
st_simpligyはトポロジー保持する関数であるが、これは個々のfeature geometrieで行われる。
つまり、ポリゴンに対して実行したものはポリゴンとなる。
しかし、2つのfeaturesが境界を共有しているときにst_simplifyを実行した場合には2つのポリゴンが同じ境界を共有しているとは限らない。これは個々のポリゴンごとに単純化が行われるためである。
sfオブジェクトに対してdplyr verbsが使えない
多くの開発者はパッケージをロードせずに、sf::のように関数の属するパッケージを明示して下記のように記述する。
i = sf::st_intersects(sf1, sf2)
しかし、sfオブジェクトに対して、dplyr::selectは利用できません。
このケースではsfパッケージをロードする必要がある。
表示されるerror/warning/messageについて
座標系は緯度経度であるが xxxは平面を仮定しています(although coordinates are longitude/latitude, xxx assumes that they are planar)
sfで利用されるgeometryに関する計算においてはGEOSライブラリを利用している。このライブラリは2次元のフラットなユクリッド距離の空間座標を考慮する。しかし、例えば極を含む緯度経度データを扱い場合にはこれだけでは不十分です。
下記に例を示すが、これは間違った結果を返すこととなる。
polygon = st_sfc( st_polygon( list(rbind(c(0, 80), c(120, 80), c(240, 80), c(0, 80)))), crs =4326) pole = st_sfc(st_point(c(0, 90)), crs = 4326) st_intersects(polygon, pole)
Sparse geometry binary predicate list of length 1, where the predicate was `intersects' 1: (empty)
st_centroidは緯度経度データに対しては正しい値を返しません(
st_centroid does not give correct centroids for longitude/latitude data)
先ほどの例と同じですが、中心は2次元空間を前提としています。
st_centroid(polygon)[[1]]
この場合中心は極である必要があります。
距離は10進数を前提としています(
dist is assumed to be in decimal degrees (arc_degrees).)
このメッセージはsfでは距離の値が度で与えられていることを示しています。このメッセージを避けるには正しいユニット(単位)を与えてやる必要があります。
pt = st_sfc(st_point(c(0,0)), crs = 4326) buf = st_buffer(polygon, 1) buf = st_buffer(polygon, units::set_units(1, degree))
両方の式のオペランドがunitオブジェクトもしくはアトミックベクトルである必要があります(
both operands of the expression should be "units" objects or _Error: $ operator is invalid for atomic vectors")
このメッセージはsfパッケージをロードした後にspatstatパッケージをロードした時に現れる。パッケージ単位で作成された単位オブジェクトの場合、unitクラスも実装するspatstatのメソッドが選択される。この問題はspatstatの後にunitパッケージをロードすることで解決できる。なお、バージョン1.53-2.012のspatstatパッケージではこれが解決されています。